● 분해(Decomposition)
- 큰 하나의 행렬을 여러 개의 작은 행렬로 분해
- 분해 과정에서 중요한 정보만 남게됨
- 라이브러리, 데이터 불러오기
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris, fetch_olivetti_faces
from sklearn.decomposition import PCA, IncrementalPCA, KernelPCA, SparsePCA
from sklearn.decomposition import TruncatedSVD, DictionaryLearning, FactorAnalysis
from sklearn.decomposition import FastICA, NMF, LatentDirichletAllocation
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
iris, labels = load_iris(return_X_y = True)
faces, _ = fetch_olivetti_faces(return_X_y = True, shuffle = True)- iris 그래프 그리기 함수
def plot_iris(iris, labels):
plt.figure()
colors = ['navy', 'purple', 'red']
for xy, label in zip(iris, labels):
plt.scatter(xy[0], xy[1], color = colors[label])- faces 그리기 함수
def show_faces(faces):
plt.figure()
# 2*3의 배열로 표현
num_rows, num_cols = 2, 3
# 총 6개씩 뜨게 됨
for i in range(num_rows * num_cols):
plt.subplot(num_rows, num_cols, i+1)
plt.imshow(np.reshape(faces[i], (64, 64)), cmap = plt.cm.gray)- 위의 함수 사용해서 그래프 그리기
plot_iris(iris[:, :2], labels)
show_faces(faces)
1. 주성분 분석(Principal Component Analysis, PCA)
- PCA를 사용해 iris 데이터 변환
- 150×4 크기의 데이터를 150×2 크기의 행렬로 압축
# 기존 iris 데이터의 shape
iris.shape
# 출력 결과
(150, 4)
# PCA 변환 후 iris 데이터의 shape
model = PCA(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# PCA로 변환한 iris 데이터 시각화
plot_iris(transformed_iris, labels)
- PCA를 통해 학습된 각 컴포넌트(6개)
- 각 컴포넌트는 얼굴의 주요 특징을 나타냄
# 기존 faces 데이터의 shape
faces.shape
# 출력 결과
(400, 4096)
# PCA 변환 후 faces 데이터의 shape
model = PCA(n_components = 6, random_state = 0)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# PCA로 변환한 faces 데이터 시각화
show_faces(faces_components)
2. Incremental PCA
- PCA는 SVD 알고리즘 실행을 위해 전체 학습용 데이터 셋을 메모리에 올려야 함
- Incremental PCA는 학습 데이터를 미니 배치 단위로 나누어 사용
- 학습 데이터가 크거나 온라인으로 PCA 적용이 필요할 때 유용
model = IncrementalPCA(n_components = 2)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)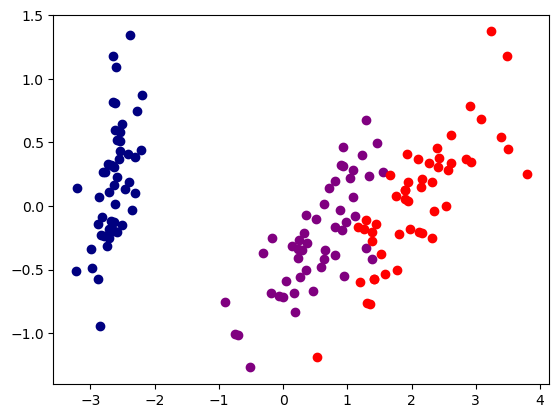
model = IncrementalPCA(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
3. Kernel PCA
- 비선형적인 형태가 Kernel로 표현될 수 있음
- 차원 축소를 위한 복잡한 비선형 투형
model = KernelPCA(n_components = 2, kernel = 'rbf', random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)
model = KernelPCA(n_components = 6)
model.fit(faces)
# Kernel PCA는 components_를 출력할 수 없어 오류가 발생함
faces_components = model.components_
4. Sparse PCA
- PCA의 주요 단점 중 하나는 주성분들이 보통 모든 입력 변수들의 선형 결합으로 나타난다는 점
- 희소 주성분 분석은 몇 개의 변수들만의 선형결합으로 주성분을 나타냄으로써 이러한 단점을 극복
model = SparsePCA(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)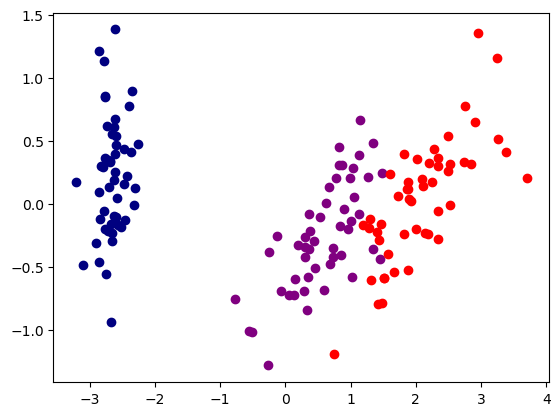
model = SparsePCA(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
5. Truncated Singular Value Decomposition(Truncated SVD)
- PCA는 정방 행렬에 대해서만 행렬 분해 가능
- SVDs는 정방 행렬 뿐만 아니라 행과 열이 다른 행렬도 분해 가능
- PCA는 밀집 행렬(Dense Matrix)에 대한 변환만 가능하지만, SVD는 희소 행렬(Sparse Matrix)에 대한 변환도 가능
- 전체 행렬 크기에 대해 Full SVD를 사용하는 경우는 적음
- 특이값이 0인 부분을 모두 제거하고 차원을 줄인 Truncated SVD를 주로 사용
model = TruncatedSVD(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)
model = TruncatedSVD(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
6. Dictionary Learning
- Sparse code를 사용하여 데이터를 가장 잘 나타내는 사전 찾기
- Sparse coding은 overcomplete 기저 벡터(basis vector)(기저보다 많은 수의 함수로 프레임 표현하는 )를 기반으로 데이터를 효율적으로 표현하기 위한 개발
- 기저 벡터는 벡터 공간에 속하는 벡터의 집합이 선형 독립이고, 다른 모든 벡터 공간의 벡터들이 그 벡터 집합의 선형 조합으로 나타남
- 이웃한 픽셀들의 가능한 모든 조합으로 이미지를 재정의
- 픽셀 정보를 넘어서 더 풍부한 표현력으로 이미지를 설명, 인식의 정확성 향상
model = DictionaryLearning(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)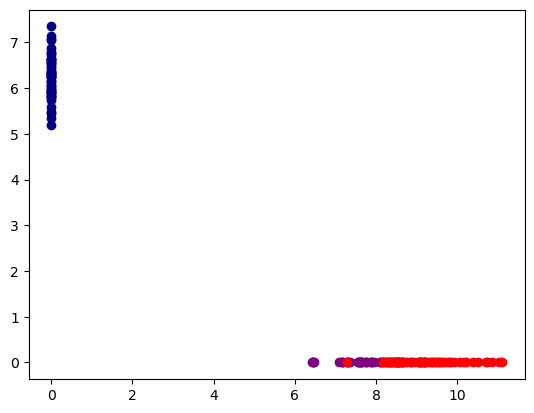
model = DictionaryLearning(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
7. Factor Analysis
- 요인 분석은 변수들 간의 상관관계를 고려하여 저변에 내재된 개념인 요인들을 추출해내는 분석방법
- 요인 분석은 변수들 간의 상관관계를 고려하여 서로 유사한 변수들끼지 묶어주는 방법
- PCA에선느 오차(error)를 고려하지 않고, 요인 분석에서는 오차(error)를 고려
model = FactorAnalysis(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)
model = FactorAnalysis(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
8. Independent Component Analysis(ICA)
- 독립 성분 분석은 다변량의 신호를 통계적으로 독립적인 하부 성분으로 분리하는 계산 방법
- ICA는 주성분을 이용하는 점은 PCA와 유사하지만, 데이터를 가장 잘 설명하는 축을 찾는 PCA와 달리 가장 독립적인 축, 독립성이 최대가 되는 벡터를 찾음
model = FastICA(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)
model = FastICA(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
9. Non-negative Matrix Factorization
- 음수 미포함 행렬 분해는 음수를 포함하지 않은 행렬 V를 음수를 포함하지 않은 행렬 W와 H의 곱으로 분해하는 알고리즘
- 숫자 5를 분해하면 2+3, 1+4 등으로 분리 가능 -> 이것과 마찬가지로 행렬을 어떤 두 개의 행렬의 곱으로 분해
model = NMF(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)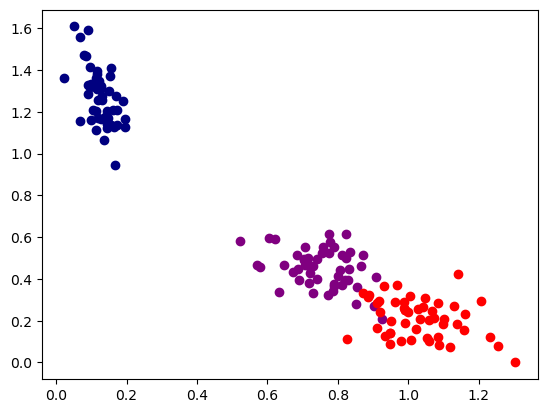
model = NMF(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
10. Latent Dirichlet Allocation(LDA)
- 잠재 디리클레 할당은 이산 자료들에 대한 확률적 생성 모형
- 디리클레 분포에 따라 잠재적인 의미 구조를 파악
- 디리클레 분포(Dirichlet distribution)는 연속 확률분포의 하나로, k차원의 실수 벡터 중 벡터의 요소가 양수이며 모든 요소를 더한 값이 1인 경우 (이를 k−1차원 단체라고 한다)에 대해 확률값이 정의되는 분포이다.
- 디리클레 분포는 베이즈 통계학에서 다항 분포에 대한 사전 켤레확률이다. 이 성질을 이용하기 위해, 디리클레 분포는 베이즈 통계학에서의 사전 확률로 자주 사용된다.
model = LatentDirichletAllocation(n_components = 2, random_state = 0)
model.fit(iris)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)
model = LatentDirichletAllocation(n_components = 6)
model.fit(faces)
faces_components = model.components_
faces_components.shape
# 출력 결과
(6, 4096)# 시각화
show_faces(faces_components)
11. Linear Discriminant Analysis(LDA)
- LDA는 PCA와 유사하게 입력 데이터 세트를 저차원 공간에 통해 차원을 축소
- LDA는 지도 학습 분류에서 사용하기 쉽도록 개별 클래스를 분별할 수 있는 기준을 최대한 유지하면서 차원 축소
- 정답이 있는 지도학습에서 사용하기 때문에 faces 데이터에 대해서는 사용 불가능
model = LinearDiscriminantAnalysis(n_components = 2)
# 정답인 labels를 같이 모델에 넣어 학습시켜 줘야함
model.fit(iris, labels)
transformed_iris = model.transform(iris)
transformed_iris.shape
# 출력 결과
(150, 2)# 시각화
plot_iris(transformed_iris, labels)
12. 압축된 표현을 사용한 학습
- 원래의 digits 데이터와 분해(decomposition)된 digits 데이터의 cross val score 비교
- digits 데이터 및 학습 모델 라이브러리 불러오기
from sklearn.neighbors import KNeighborsClassifier
from sklearn.svm import SVC
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import load_digits
from sklearn.model_selection import cross_val_score
# min_max 스케일링 하는 함수
def min_max_scale(x):
min_value, max_value = np.min(x, 0), np.max(x, 0)
x = (x - min_value) / (max_value - min_value)
return x
# digits 데이터 그래프 그리는 함수
def plot_digits(digits, labels):
digits = min_max_scale(digits)
ax = plt.subplot(111, projection = '3d')
for i in range(digits.shape[0]):
ax.text(digits[i, 0], digits[i, 1], digits[i, 2],
str(labels[i]), color = plt.cm.Set1(labels[i] / 10.),
fontdict = {'weight': 'bold', 'size': 9})
ax.view_init(4, -72)
# digits 데이터 불러온 뒤 NMF로 분해
# 기존 데이터와 분해된 데이터 shape 비교
digits = load_digits()
nmf = NMF(n_components = 3)
nmf.fit(digits.data)
decomposed_digits = nmf.transform(digits.data)
print(digits.data.shape)
print(decomposed_digits.shape)
print(decomposed_digits)
# 출력 결과
(1797, 64)
(1797, 3)
[[0.48392621 0. 1.24523912]
[0.5829615 1.4676756 0.07150889]
[0.61515882 1.10963207 0.387782 ]
...
[0.55272665 1.26056519 0.72094739]
[0.7872562 0.2789873 1.04952028]
[0.78507412 0.67250884 0.92677982]]# 시각화
plt.figure(figsize = (20, 10))
plot_digits(decomposed_digits, digits.target)
- KNN
# 분해 전
knn = KNeighborsClassifier()
score = cross_val_score(
estimator = knn,
X = digits.data, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.94722222 0.95555556 0.96657382 0.98050139 0.9637883 ]
mean cross val score: 0.9627282575054161 (+/- 0.011168537355954218)
# 분해 후
knn = KNeighborsClassifier()
score = cross_val_score(
estimator = knn,
X = decomposed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.54722222 0.58055556 0.64066852 0.59610028 0.56267409]
mean cross val score: 0.5854441349427422 (+/- 0.03214521445075084)
- SVC
# 분해 전
svm = SVC()
score = cross_val_score(
estimator = svm,
X = digits.data, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.96111111 0.94444444 0.98328691 0.98885794 0.93871866]
mean cross val score: 0.9632838130609718 (+/- 0.02008605863225686)
# 분해 후
svm = SVC()
score = cross_val_score(
estimator = svm,
X = decomposed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.61388889 0.62222222 0.66016713 0.60167131 0.59888579]
mean cross val score: 0.6193670690188796 (+/- 0.022070024720937543)
- Decision Tree
# 분해 전
decision_tree = DecisionTreeClassifier()
score = cross_val_score(
estimator = decision_tree,
X = digits.data, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.78333333 0.69722222 0.78830084 0.83286908 0.78830084]
mean cross val score: 0.7780052615289385 (+/- 0.04421837659784472)
# 분해 후
decision_tree = DecisionTreeClassifier()
score = cross_val_score(
estimator = decision_tree,
X = decomposed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.57222222 0.50833333 0.57938719 0.5821727 0.52924791]
mean cross val score: 0.5542726709996905 (+/- 0.0298931375955385)
- Random Forest
# 분해 전
random_forest = RandomForestClassifier()
score = cross_val_score(
estimator = random_forest,
X = digits.data, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.93888889 0.90555556 0.97214485 0.96657382 0.91086351]
mean cross val score: 0.9388053234292789 (+/- 0.02745509790638632)
# 분해 후
random_forest = RandomForestClassifier()
score = cross_val_score(
estimator = random_forest,
X = decomposed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.58888889 0.60277778 0.64066852 0.59052925 0.54874652]
mean cross val score: 0.594322191272052 (+/- 0.029463634023029848)
- 전반적으로 분해한 데이터에서 성능이 떨어진 모습
13. 복원된 표현을 사용한 학습
- 분해 후 복원된 행렬을 사용해 학습
- 데이터 행렬 복원
# 분해된 행렬에 곱하기 연산을 통해 원래의 행렬로 복원
components = nmf.components_
reconstructed_digits = decomposed_digits @ components
print(digits.data.shape)
print(decomposed_digits.shape)
print(reconstructed_digits.shape)
# 출력 결과
(1797, 64)
(1797, 3)
(1797, 64)# reconstructed digits 시각화
plt.figure(figsize = (16, 8))
plt.suptitle('Re-Constructed digits')
for i in range(10):
plt.subplot(2, 5, i+1)
plt.xticks([])
plt.yticks([])
plt.imshow(reconstructed_digits[i].reshape(8, 8))
- KNN
knn = KNeighborsClassifier()
score = cross_val_score(
estimator = knn,
X = reconstructed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.54166667 0.59444444 0.66295265 0.57660167 0.57381616]
mean cross val score: 0.5898963169297431 (+/- 0.04029722337499952)
- SVM
svm = SVC()
score = cross_val_score(
estimator = svm,
X = reconstructed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.62777778 0.60555556 0.66016713 0.61002786 0.5821727 ]
mean cross val score: 0.6171402042711235 (+/- 0.025969174809053776)
- Decision Tree
decision_tree = DecisionTreeClassifier()
score = cross_val_score(
estimator = decision_tree,
X = reconstructed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.57777778 0.51666667 0.53481894 0.56824513 0.55153203]
mean cross val score: 0.5498081089445992 (+/- 0.0221279380012718)
- Random Forest
random_forest = RandomForestClassifier()
score = cross_val_score(
estimator = random_forest,
X = reconstructed_digits, y = digits.target,
cv = 5
)
print(score)
print('mean cross val score: {} (+/- {})'.format(score.mean(), score.std()))
# 출력 결과
[0.58055556 0.55833333 0.65181058 0.59610028 0.57660167]
mean cross val score: 0.592680284741566 (+/- 0.031916555892366645)
- 새로 복원한 데이터에서도 큰 성능 향상점은 없음
14. 이미지 복원
# faces 데이터를 train와 test 데이터로 분리
from sklearn.model_selection import train_test_split
train_faces, test_faces = train_test_split(faces, test_size = 0.1)
show_faces(train_faces)
show_faces(test_faces)
# 테스트 데이터에는 랜덤한 점에 0값을 주어 검은색의 점으로 노이즈를 생성
damaged_faces = []
for face in test_faces:
idx = np.random.choice(range(64 * 64), size = 1024)
damaged_face = face.copy()
damaged_face[idx] = 0.
damaged_faces.append(damaged_face)
show_faces(damaged_faces)
# train 데이터로 NMF 분해 학습
nmf = NMF(n_components = 10)
nmf.fit(train_faces)# 노이즈를 준 test 데이터를 NMF 모델에 넣어 분해하고 다시 복원하는 과정으로 노이즈 제거
# damaged_faces의 형식을 float32로 변환하는 과정 필요
damaged_faces = np.asarray(damaged_faces, dtype = np.float32)
matrix1 = nmf.transform(damaged_faces)
matrix2 = nmf.components_
show_faces(matrix1 @ matrix2)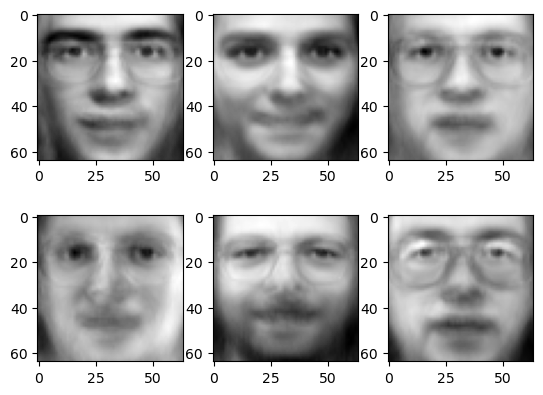
# 분해 components를 조절하여 정교함 조절 가능, 더 높은 수로 분해할수록 더 정교하게 복원할 수 있음
nmf = NMF(n_components = 100)
nmf.fit(train_faces)
matrix1 = nmf.transform(damaged_faces)
matrix2 = nmf.components_
show_faces(matrix1 @ matrix2)
nmf = NMF(n_components = 300)
nmf.fit(train_faces)
matrix1 = nmf.transform(damaged_faces)
matrix2 = nmf.components_
show_faces(matrix1 @ matrix2)
'Python > Machine Learning' 카테고리의 다른 글
| 머신러닝 기본 과정(Dacon, Kaggle 분류 및 회귀분석 초기 EDA 및 Feature Engineering 용) (0) | 2023.09.03 |
|---|---|
| [머신러닝 알고리즘] 추천 시스템 (1) | 2023.03.03 |
| [머신러닝 알고리즘] 다양체 학습 (0) | 2023.03.03 |
| [머신러닝 알고리즘] 군집화 (1) | 2023.03.02 |
| [머신러닝 알고리즘] XGBoost, LightGBM (0) | 2023.02.23 |