● XGBoost
- 트리 기반의 앙상블 기법
- 분류에 있어서 다른 알고리즘보다 좋은 예측 성능을 보여줌
- XGBoost는 GBM 기반이지만, GBM의 단점인 느린 수행 시간과 과적합 규제 부재 등의 문제를 해결
- 병렬 CPU 환경에서 빠르게 학습 가능
- 필요 라이브러리
from sklearn.datasets import load_iris, load_breast_cancer, load_wine, load_diabetes
from sklearn.model_selection import train_test_split, cross_validate
from sklearn.metrics import accuracy_score, precision_score, recall_score
import xgboost as xgb
from xgboost import XGBClassifier, XGBRegressor
from xgboost import plot_importance, plot_tree
import graphviz
import matplotlib.pyplot as plt
1. 파이썬 기반 XGBoost
- 유방암 데이터로 연습
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, test_size = 0.2, random_state = 42)
# XGBoost는 XGBoost만의 데이터 형식인 DMatrix 형식을 사용하므로 다음과 같이 train과 test 데이터를 변환
dtrain = xgb.DMatrix(data = X_train, label = y_train)
dtest = xgb.DMatrix(data = X_test, label = y_test)
# 파라미터
params = {
'max_depth': 3,
'eta': 0.1,
'objective': 'binary:logistic',
'eval_metric': 'logloss',
'early_stopping': 100
}
num_rounds = 400
# 모델 생성
evals = [(dtrain, 'train'), (dtest, 'eval')]
xgb_model = xgb.train(params = params, dtrain = dtrain, num_boost_round = num_rounds, early_stopping_rounds = 100, evals = evals)
- xgb_model 생성 결과, logloss가 더 이상 감소되지 않을 때까지 모델 최적화
- 계속 감소한다면 사전에 정해둔 round 횟수(num_rounds = 400)만큼만 진행한 뒤 모델 생성 종료
- 가장 마지막 모델로 생성

- train 데이터로 생성한 모델에 test 데이터를 넣어 예측값 출력(상위 10개만)
import numpy as np
predicts = xgb_model.predict(dtest)
print(np.round(predicts[:10], 3))
- 정확도, 정밀도, 재현율 출력
# 데이터 생성 결과를 이원화(0 또는 1로)하여 2*2 테이블 상에서 정확도, 정밀도, 재현율을 계산할 수 있도록 변경
preds = [1 if x > 0.5 else 0 for x in predicts]
print(preds[:10])
print("정확도: {}".format(accuracy_score(y_test, preds)))
print("정밀도: {}".format(precision_score(y_test, preds)))
print("재현율: {}".format(recall_score(y_test, preds)))
# 출력 결과
정확도: 0.9736842105263158
정밀도: 0.9722222222222222
재현율: 0.9859154929577465
- 변수들의 중요도를 알아보기 위한 그래프 출력
fig, ax = plt.subplots(figsize = (10, 12))
plot_importance(xgb_model, ax = ax)
- xgb 모델을 트리 형식으로 표현한 그래프 출력
dot_data = xgb.to_graphviz(xgb_model)
dot_data
2. XGBClassifier
- 붓꽃 데이터
iris = load_iris()
X_train, X_test, y_train, y_test = train_test_split(iris.data, iris.target, test_size = 0.2, random_state = 42)- XGBClassifier 모델 생성
xgbc = XGBClassifier(n_estimators = 400, learning_rate = 0.1, max_depth = 3)
xgbc.fit(X_train, y_train)
preds = xgbc.predict(X_test)
preds_proba = xgbc.predict_proba(X_test)[:, 1]
cross_val = cross_validate(
estimator = xgbc,
X = iris.data, y = iris.target,
cv = 5
)
print('avg fit time: {} (+/- {})'.format(cross_val['fit_time'].mean(), cross_val['fit_time'].std()))
print('avg score time: {} (+/- {})'.format(cross_val['score_time'].mean(), cross_val['score_time'].std()))
print('avg test score: {} (+/- {})'.format(cross_val['test_score'].mean(), cross_val['test_score'].std()))
# 출력 결과
avg fit time: 0.38040671348571775 (+/- 0.12548339326469174)
avg score time: 0.0038000106811523437 (+/- 0.001165665213187403)
avg test score: 0.96 (+/- 0.024944382578492935)- 모델에서 데이터의 각 변수 중요도 출력
fig, ax = plt.subplots(figsize = (10, 12))
plot_importance(xgbc, ax = ax)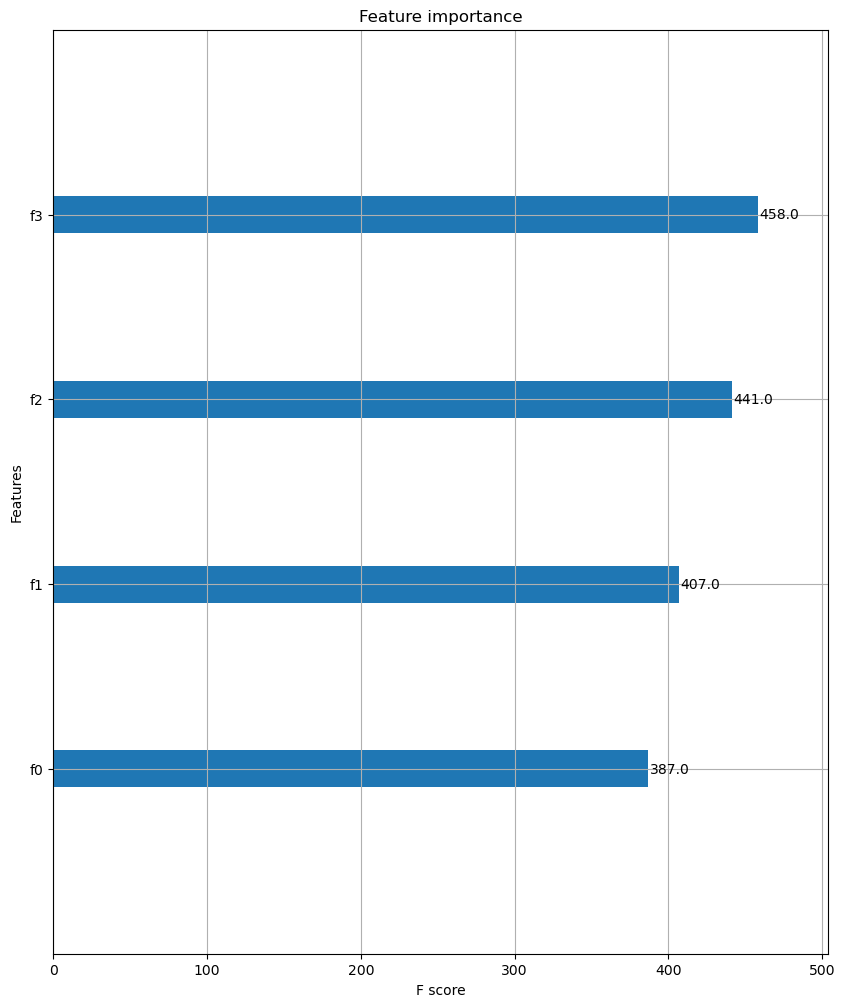
- xgb 모델을 트리 형식으로 출력
dot_data = xgb.to_graphviz(xgbc)
dot_data
- 와인 데이터
wine = load_wine()
X_train, X_test, y_train, y_test = train_test_split(wine.data, wine.target, test_size = 0.2, random_state = 42)- XGBClassifier 모델 생성
- 파라미터의 종류
- n_estimators : 학습 모델의 수, 많아질수록 성능 향상의 가능성이 있으나, 속도가 느려짐
- learning_rate : 학습률, 너무 크면 gradient 발산의 가능성이 있으며, 너무 작으면 학습이 느림
- max_depth : 최대 탐색 깊이, 너무 크면 과적합의 가능성, 너무 작으면 학습 성능 저하
- min_samples_split : 분할 종료 최소 샘플 수, 큰 수면 과적합을 막지만 학습 성능 저하 가능성
- min_samples_leaf : leaf node가 되기 위한 최소 샘플 수, min_samples_split과 비슷한 용도
xgbc = XGBClassifier(n_estimators = 400, learning_rate = 0.1, max_depth = 3)
xgbc.fit(X_train, y_train)
preds = xgbc.predict(X_test)
preds_proba = xgbc.predict_proba(X_test)[:, 1]
cross_val = cross_validate(
estimator = xgbc,
X = iris.data, y = iris.target,
cv = 5
)
print('avg fit time: {} (+/- {})'.format(cross_val['fit_time'].mean(), cross_val['fit_time'].std()))
print('avg score time: {} (+/- {})'.format(cross_val['score_time'].mean(), cross_val['score_time'].std()))
print('avg test score: {} (+/- {})'.format(cross_val['test_score'].mean(), cross_val['test_score'].std()))
# 출력 결과
avg fit time: 0.8099905490875244 (+/- 0.2795799385941016)
avg score time: 0.004575538635253906 (+/- 0.0016525575848722433)
avg test score: 0.96 (+/- 0.024944382578492935)- 모델에서 데이터의 각 변수 중요도 출력
fig, ax = plt.subplots(figsize = (10, 12))
plot_importance(xgbc, ax = ax)
- xgb 모델을 트리 형식으로 출력
dot_data = xgb.to_graphviz(xgbc)
dot_data
3. XGBRegressor
- XGB는 Classifier에 더 특화되어 있지만 Regressor도 할 수는 있음
- 당뇨병 데이터
diabetes = load_diabetes()
X_train, X_test, y_train, y_test = train_test_split(diabetes.data, diabetes.target, test_size = 0.2, random_state = 42)- XGBRegressor 모델 생성
- 파라미터의 종류
- n_estimators : 학습 모델의 수, 많아질수록 성능 향상의 가능성이 있으나, 속도가 느려짐
- learning_rate : 학습률, 너무 크면 gradient 발산의 가능성이 있으며, 너무 작으면 학습이 느림
- max_depth : 최대 탐색 깊이, 너무 크면 과적합의 가능성, 너무 작으면 학습 성능 저하
- min_samples_split : 분할 종료 최소 샘플 수, 큰 수면 과적합을 막지만 학습 성능 저하 가능성
- min_samples_leaf : leaf node가 되기 위한 최소 샘플 수, min_samples_split과 비슷한 용도
- objective : 목적함수, reg:linear(linear-regression), binary:logistic(binary-logistic-classification), count:poisson(count data poison regression) 등 다양
- XGBClassifier보다 test score가 낮게 나
xgbr = XGBRegressor(n_estimators = 400, learning_rate = 0.1, max_depth = 3, objective = 'reg:squarederror')
xgbr.fit(X_train, y_train)
preds = xgbr.predict(X_test)
cross_val = cross_validate(
estimator = xgbr,
X = diabetes.data, y = diabetes.target,
cv = 5
)
print('avg fit time: {} (+/- {})'.format(cross_val['fit_time'].mean(), cross_val['fit_time'].std()))
print('avg score time: {} (+/- {})'.format(cross_val['score_time'].mean(), cross_val['score_time'].std()))
print('avg test score: {} (+/- {})'.format(cross_val['test_score'].mean(), cross_val['test_score'].std()))
# 출력 결과
avg fit time: 0.3746964454650879 (+/- 0.027832593554179098)
avg score time: 0.005658674240112305 (+/- 0.0017620359721342335)
avg test score: 0.30005291115066424 (+/- 0.07589309667544569)- 모델에서 데이터의 각 변수 중요도 출력
fig, ax = plt.subplots(figsize = (10, 12))
plot_importance(xgbr, ax = ax)
- xgb 모델을 트리 형식으로 출력
dot_data = xgb.to_graphviz(xgbr)
dot_data
● LightGBM
- XGBoost보다 2년쯤 뒤에 나와 더 개선됨
- 빠른 학습과 예측 시간
- 더 적은 메모리 사용
- 범주형 특징의 자동 변환과 최적 분할
- 필요 라이브러리
from lightgbm import LGBMClassifier, LGBMRegressor
from lightgbm import plot_importance, plot_metric, plot_tree
1. LGBMClassifier
- 붓꽃 데이터
iris = load_iris()
X_train, X_test, y_train, y_test = train_test_split(iris.data, iris.target, test_size = 0.2, random_state = 42)- LGBMClassifier 모델 생성
lgbmc = LGBMClassifier(n_estimators = 400)
evals = [(X_test, y_test)]
lgbmc.fit(X_train, y_train, early_stopping_rounds = 100, eval_metric = 'logloss', eval_set = evals, verbose = True)
preds = lgbmc.predict(X_test)
cross_val = cross_validate(
estimator = lgbmc,
X = iris.data, y = iris.target,
cv = 5
)
print('avg fit time: {} (+/- {})'.format(cross_val['fit_time'].mean(), cross_val['fit_time'].std()))
print('avg score time: {} (+/- {})'.format(cross_val['score_time'].mean(), cross_val['score_time'].std()))
print('avg test score: {} (+/- {})'.format(cross_val['test_score'].mean(), cross_val['test_score'].std()))
# 출력 결과
avg fit time: 0.19687752723693847 (+/- 0.08293570240505971)
avg score time: 0.001802968978881836 (+/- 0.0004004494069017428)
avg test score: 0.9600000000000002 (+/- 0.04898979485566355)- lgbm 모델이 실행되며 logloss의 변화를 관찰할 수 있는 그래프 출력
- 성능이 어떻게 향상되고 있는지 볼 수 있음
plot_metric(lgbmc)
- 변수 중요도
- LGBM 전용 plot_importance 사용
plot_importance(lgbmc, figsize = (10, 12))
- 트리 형태로 출력
plot_tree(lgbmc, figsize = (28, 24))
- 와인 데이터
wine = load_wine()
X_train, X_test, y_train, y_test = train_test_split(wine.data, wine.target, test_size = 0.2, random_state = 42)- LGBMClassifier 모델 생성
lgbmc = LGBMClassifier(n_estimators = 400)
evals = [(X_test, y_test)]
lgbmc.fit(X_train, y_train, early_stopping_rounds = 100, eval_metric = 'logloss', eval_set = evals, verbose = True)
preds = lgbmc.predict(X_test)
cross_val = cross_validate(
estimator = lgbmc,
X = wine.data, y = wine.target,
cv = 5
)
print('avg fit time: {} (+/- {})'.format(cross_val['fit_time'].mean(), cross_val['fit_time'].std()))
print('avg score time: {} (+/- {})'.format(cross_val['score_time'].mean(), cross_val['score_time'].std()))
print('avg test score: {} (+/- {})'.format(cross_val['test_score'].mean(), cross_val['test_score'].std()))
# 출력 결과
avg fit time: 0.6419896125793457 (+/- 0.21000223475863156)
avg score time: 0.003860330581665039 (+/- 0.002245084936532545)
avg test score: 0.9776190476190475 (+/- 0.01119469694127331)- lgbm 모델이 실행되며 logloss의 변화를 관찰할 수 있는 그래프 출력
plot_metric(lgbmc)
- 변수 중요도
plot_importance(lgbmc, figsize = (10, 12))
- 트리 형태로 출력
plot_tree(lgbmc, figsize = (28, 24))
- 유방암 데이터
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, test_size = 0.2, random_state = 42)- LGBMClassifier 모델 생성
lgbmc = LGBMClassifier(n_estimators = 400)
evals = [(X_test, y_test)]
lgbmc.fit(X_train, y_train, early_stopping_rounds = 100, eval_metric = 'logloss', eval_set = evals, verbose = True)
preds = lgbmc.predict(X_test)
cross_val = cross_validate(
estimator = lgbmc,
X = cancer.data, y = cancer.target,
cv = 5
)
print('avg fit time: {} (+/- {})'.format(cross_val['fit_time'].mean(), cross_val['fit_time'].std()))
print('avg score time: {} (+/- {})'.format(cross_val['score_time'].mean(), cross_val['score_time'].std()))
print('avg test score: {} (+/- {})'.format(cross_val['test_score'].mean(), cross_val['test_score'].std()))
# 출력 결과
avg fit time: 1.5169551372528076 (+/- 0.7215118218596442)
avg score time: 0.0062160491943359375 (+/- 0.002043592719798801)
avg test score: 0.9736531594472908 (+/- 0.015674460437800138)- lgbm 모델이 실행되며 logloss의 변화를 관찰할 수 있는 그래프 출력
plot_metric(lgbmc)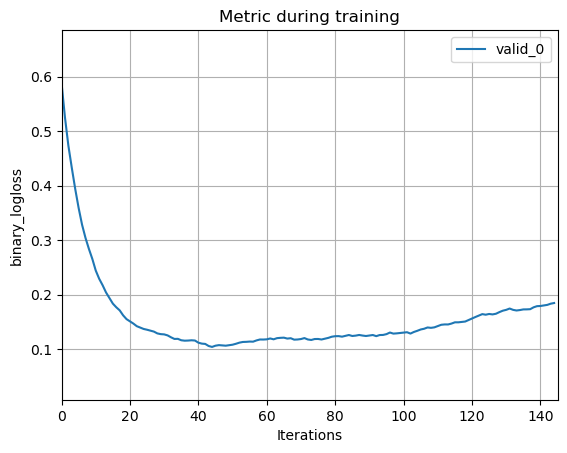
- 변수 중요도
plot_importance(lgbmc, figsize = (10, 12))
- 트리 형태로 출력
plot_tree(lgbmc, figsize = (28, 24))
2. LGBMRegressor
- 당뇨병 데이터
diabetes = load_diabetes()
X_train, X_test, y_train, y_test = train_test_split(diabetes.data, diabetes.target, test_size = 0.2, random_state = 42)- LGBMRegressor 모델 생성
lgbmr = LGBMRegressor(n_estimators = 400)
evals = [(X_test, y_test)]
lgbmr.fit(X_train, y_train, early_stopping_rounds = 100, eval_metric = 'logloss', eval_set = evals, verbose = True)
preds = lgbmr.predict(X_test)
cross_val = cross_validate(
estimator = lgbmr,
X = diabetes.data, y = diabetes.target,
cv = 5
)
print('avg fit time: {} (+/- {})'.format(cross_val['fit_time'].mean(), cross_val['fit_time'].std()))
print('avg score time: {} (+/- {})'.format(cross_val['score_time'].mean(), cross_val['score_time'].std()))
print('avg test score: {} (+/- {})'.format(cross_val['test_score'].mean(), cross_val['test_score'].std()))
# 출력 결과
avg fit time: 1.210813045501709 (+/- 0.6173186047501074)
avg score time: 0.010993432998657227 (+/- 0.009632501449539222)
avg test score: 0.30867643947179507 (+/- 0.07010708786960605)- lgbm 모델이 실행되며 logloss의 변화를 관찰할 수 있는 그래프 출력
plot_metric(lgbmr)
- 변수 중요도
plot_importance(lgbmr, figsize = (10, 12))
- 트리 형태로 출력
plot_tree(lgbmr, figsize = (28, 24))
'Python > Machine Learning' 카테고리의 다른 글
| [머신러닝 알고리즘] 다양체 학습 (0) | 2023.03.03 |
|---|---|
| [머신러닝 알고리즘] 군집화 (1) | 2023.03.02 |
| [머신러닝 알고리즘] 앙상블 (0) | 2023.02.18 |
| [머신러닝 알고리즘] 결정 트리 (0) | 2023.01.18 |
| [머신러닝 알고리즘] 서포트 벡터 머신 (0) | 2023.01.13 |